北京时间2024年3月28日,中国科学院分子植物科学卓越创新中心/中国科学院合成生物学重点实验室张余研究组在国际著名学术期刊nature上发表题为“structural basis of exoribonuclease-mediated mrna transcription termination”的研究论文,该研究揭示了酵母mrna转录终止的分子机制。

研究背景
转录作为中心法则中必不可少的一步,在基因表达过程中承担重要作用。转录按照其反应过程可以分为转录起始,转录延伸,转录终止。近年来转录起始和转录延伸的机制逐渐被揭开。然而,由于转录终止的动态和不稳定特性,其机制仍不清楚。转录终止(transcription termination)是指rna聚合酶(rna polymerase,rnap)停止rna延伸、释放rna并从dna解离的过程。正确的转录终止对mrna的稳定性和rna聚合酶的高效循环利用至关重要。酵母细胞rna聚合酶ii(rna polymerase ii,pol ii)的转录终止机制根据其合成的rna类型主要分为两类,第一类是mrna的转录终止,第二类是non-coding rna的转录终止。
mrna的转录终止机制在真核生物中普遍保守。当pol ii转录至基因末端的多聚腺苷化信号(polyadenylation site; pas)时,pre-mrna在pas位点下游被切割分为两段。切割后的mrna完成polya加尾后被转运至细胞质翻译,而pol ii仍然朝下游转录继续合成rna,其转录终止依赖一个保守的5’-3’ rna外切酶(酵母rat1,哺乳动物xrn2,植物xrn3),针对真核细胞mrna转录终止的机制已经被研究了多年,提出了包括“鱼雷”、“异构”等多个模型,但是其机制尚存争议。异构模型认为,核酸外切酶rat1结合诱导pol ii构象变化,促进mrna自发解离。鱼雷模型认为,核酸外切酶rat1切割下游rna并追赶pol ii,当rat1赶上pol ii后触发mrna解离。转录终止具体通过何种机制尚存争论。
研究内容
鉴于mrna转录终止过程快速连续,难以捕捉其中间状态。研究团队利用磷硫键修饰的不同长度rna组装pol ii转录终止复合物,重构了外切酶缩短rna的中间态过程,并解析了上述中间状态的复合物结构(图1)。研究团队发现外切酶稳定结合在pol ii的rna通道外侧,rna从pol ii的活性中心延伸到了外切酶的活性中心。外切酶的结合位置和pol ii的转录延伸因子互相重叠,外切酶结合促使了延伸因子的解离,解释了外切酶延缓pol ii转录延伸速率的原因。根据以上结构信息,研究团队提出了外切酶介导的mrna转录终止机制:外切酶覆盖rna通道出口,直接将pol ii转录的rna引导至催化中心,利用其rna外切酶和移位酶缩短rna长度的同时,对mrna产生牵引力从而将其从pol ii 催化中心拖拽出来,最终解离mrna并终止pol ii 转录(图2)。研究团队的工作表明mrna的转录终止符合修正版的“鱼雷”模型,pol ii类似航行的军舰、外切酶鱼雷追赶上pol ii之后,吸附在pol ii上,随后利用其水解rna转化的机械力将rna从pol ii中拖拽出来。
比较细菌、古菌和真核生物mrna转录终止,研究团队提出三域生物利用相似的方式终止mrna合成。这些终止因子均结合在聚合酶的rna通道外侧,真核生物利用rat1/xrn2/xrn3的外切酶活性解离mrna,细菌利用rho的rna移位酶活性解离mrna,古菌利用ftta的外切酶活性解离mrna。
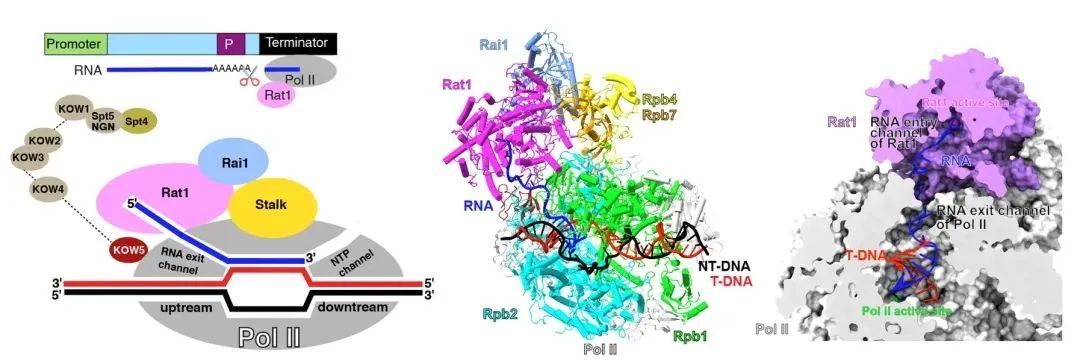
图1 外切酶rat1与pol ii形成的复合物结构
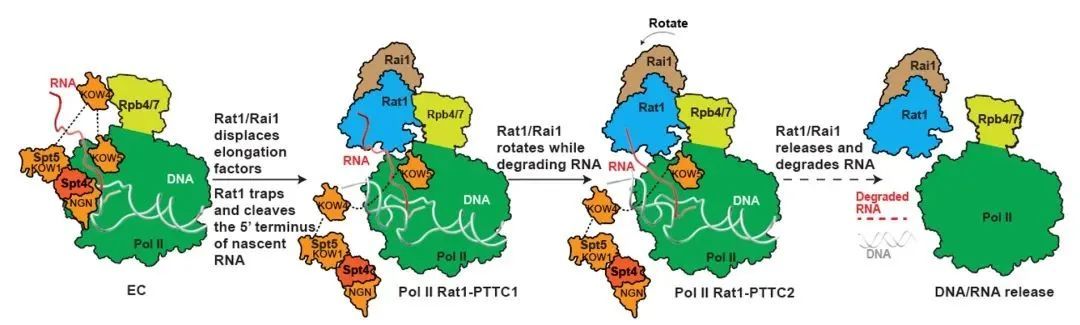
图2 外切酶rat1介导的转录终止模型
相关信息
中国科学院分子植物科学卓越创新中心张余组博士生曾媛(已毕业)为论文的第一作者,张余研究员为通讯作者。

张余研究员(左)与曾媛博士

论文详情请点击左下角“阅读原文”
来源:中国科学院分子植物科学卓越创新中心
编辑:拾
↓分享
↓点赞
↓在看
上观号作者:上海科技
